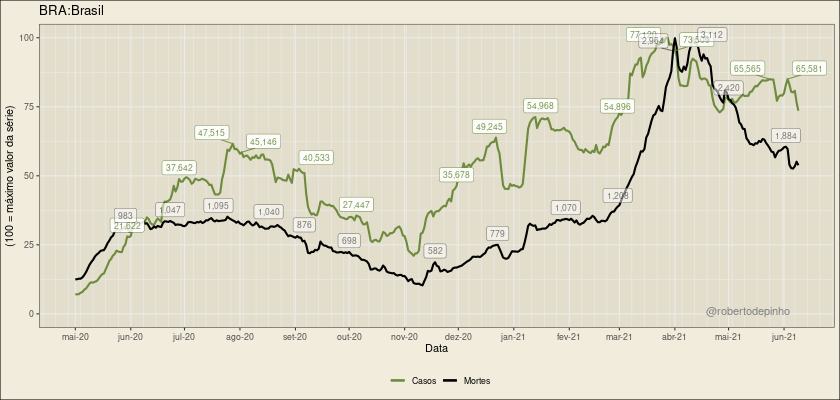
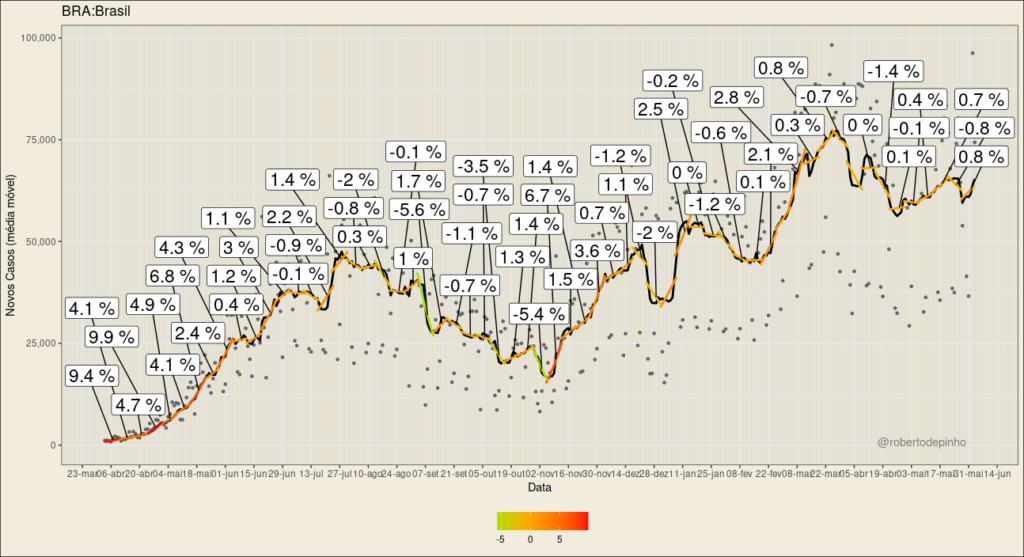
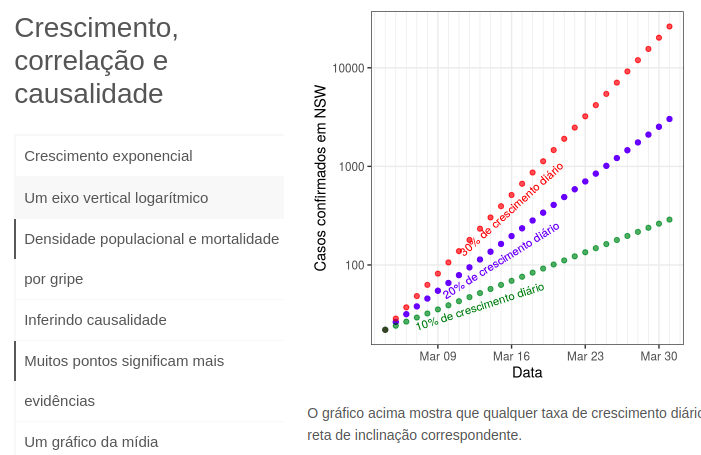
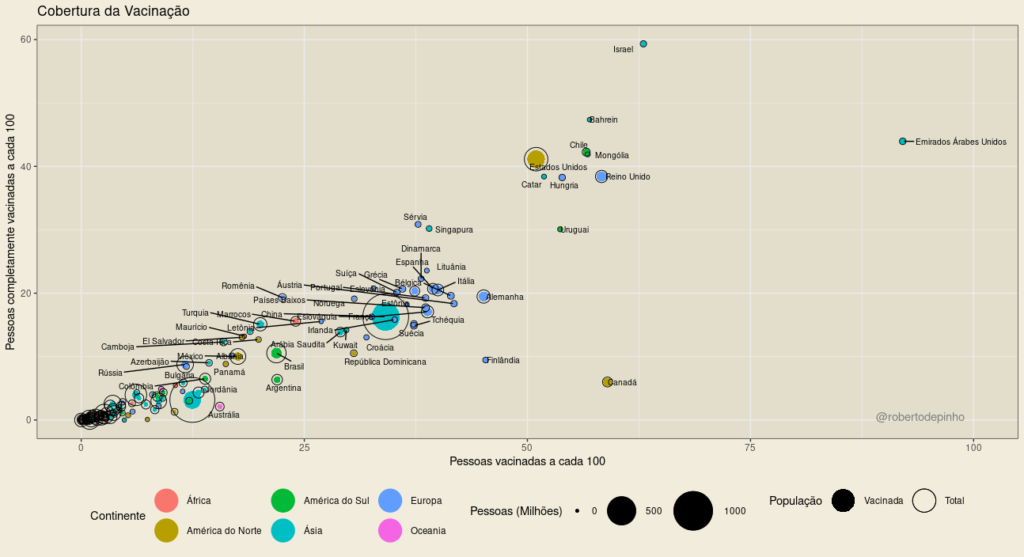
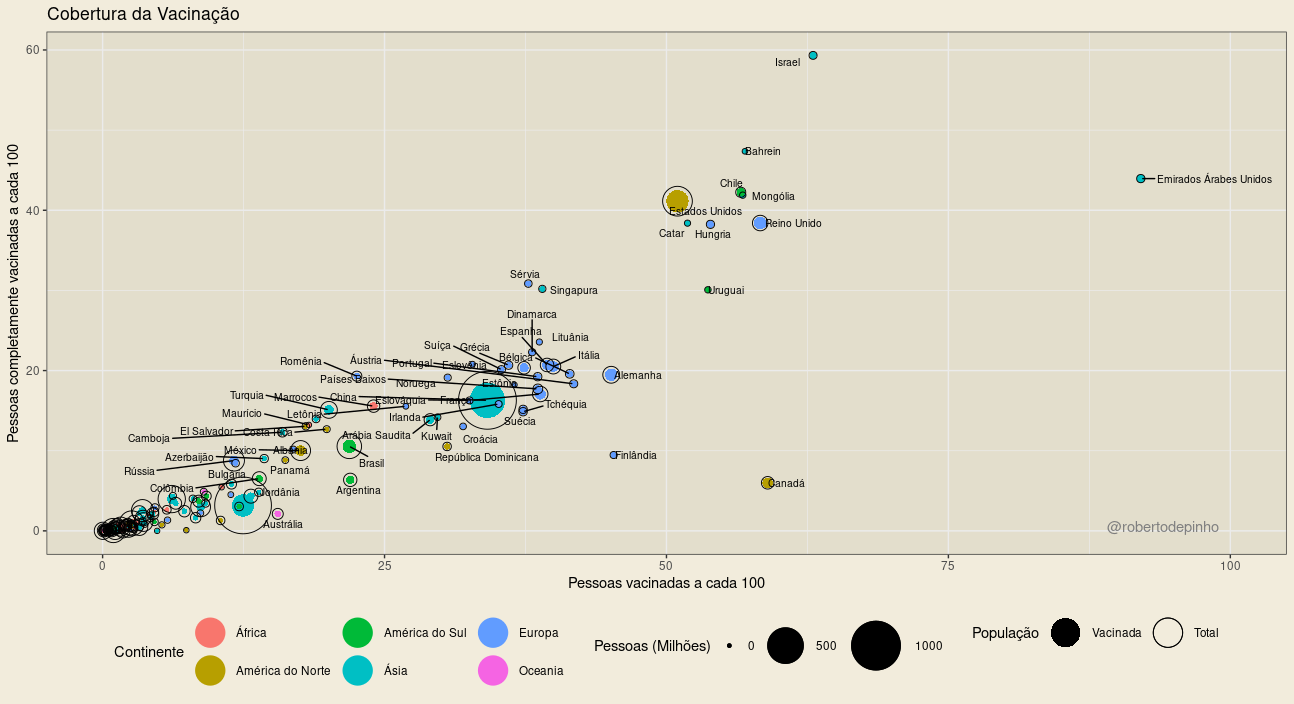
No gráfico acima, cada ponto representa um país. O tamanho do círculo externo em preto representa a sua população total. O círculo interno, colorido, representa a população vacinada (que recebeu ao menos uma dose), em números absolutos.
A fonte dos dados é o Our World in Data. Nesta fonte, nem todos os países possuem do dado da população vacinada, apenas do número de doses administradas. Nestes casos, regressão linear entre a razão de vacinas e população e, respectivamente, a população vacinada com ao menos uma dose e a população com vacinação completa é utilizada para estimativa. São utilizados os dados do último dia disponível de cada país. Exibidos apenas países com mais de 1 milhão de habitantes.
Gráficos postados diariamente no Twitter em @robertodepinho
Outros gráficos disponíveis em: Gráficos Covid-19
Código em R*:
vaccminder <- function(lang = "pt"){
#column selection
selected_columns = c("iso_code", "location" ,"continent", "date", "population","total_vaccinations", "people_fully_vaccinated", "people_vaccinated")
#read data
df = read.csv("https://raw.githubusercontent.com/owid/covid-19-data/master/public/data/owid-covid-data.csv", stringsAsFactors = F)
df = subset(df, !is.na(total_vaccinations))
#keep only last day for each country
last_date = aggregate(date ~ iso_code, df, max)
colnames(last_date)[2] = "last_date"
df = merge(df, last_date, by = "iso_code", all.x = T)
df_ld = df[df$date == df$last_date, selected_columns]
#calculation
df_ld$people_vacc_ratio = df_ld$people_vaccinated / df_ld$total_vaccinations
median_ratio = median(df_ld$people_vacc_ratio, na.rm=T)
df_ld$people_fully_vacc_ratio = df_ld$people_fully_vaccinated / df_ld$total_vaccinations
median_ratio_fully = median(df_ld$people_fully_vacc_ratio, na.rm=T)
df_ld$people_vaccinated_est = ifelse(!is.na(df_ld$people_vaccinated),
df_ld$people_vaccinated,
df_ld$total_vaccinations * median_ratio)
df_ld$people_fully_vaccinated_est = ifelse(!is.na(df_ld$people_fully_vaccinated),
df_ld$people_fully_vaccinated,
df_ld$total_vaccinations * median_ratio_fully)
df_ld$non_vacc = df_ld$population - df_ld$people_vaccinated_est
df_ld$people_vaccinated_per_hundred = df_ld$people_vaccinated_est / df_ld$population * 100
df_ld$people_fully_vaccinated_per_hundred = df_ld$people_fully_vaccinated_est / df_ld$population * 100
#translation list & df
translation <- list(
"title" = list("en" = "Vaccination Coverage", "pt"="Cobertura da Vacinação"),
"people_1M" = list("en" = "People (millions)", "pt"="Pessoas (Milhões)"),
"continent" = list("en" = "Continent", "pt"="Continente"),
"vacc" = list("en" = "Vaccinated", "pt"="Vacinada"),
"tot" = list("en" = "Total", "pt"="Total"),
"pop" = list("en" = "Population", "pt"="População"),
"vacc_100" = list("en" = "People vaccinated per hundred", "pt"="Pessoas vacinadas a cada 100"),
"vacc_full_100" = list("en" = "People fully vaccinated per hundred", "pt"="Pessoas completamente vacinadas a cada 100")
)
continents = data.frame(continent = c("Asia", "Africa", "Europe", "South America", "Oceania", "North America"),
continent_pt = c("Ásia", "África", "Europa", "América do Sul", "Oceania", "América do Norte"))
#translate country names
library(countrycode)
df_ld$country_name = countrycode(df_ld$iso_code, origin = "iso3c", destination = paste("cldr.name.", lang, sep=""))
df_ld = merge(df_ld, continents, by = "continent", all.x = T )
if(lang == "en"){
df_ld$continent_name = df_ld$continent
} else {
df_ld$continent_name = df_ld$continent_pt
}
#chart
library(ggplot2)
library(scales)
library(ggrepel)
source("src/theme_black.R")
#select chart data
dfw = df_ld
dfw = dfw[ substr(dfw$iso_code, 1,4) != "OWID",]
dfw = dfw[ dfw$population> 10^6,]
covidPlot = ggplot(data=dfw)
covidPlot = covidPlot + ggtitle(translation[["title"]][[lang]])
covidPlot = covidPlot + aes(x=people_vaccinated_per_hundred, y=people_fully_vaccinated_per_hundred, size = population/10^6,
label = country_name)
covidPlot = covidPlot + annotate("text", label = "@robertodepinho", color= "grey50",
x = 100, y = 0, vjust=0.1, hjust=1.1)
covidPlot = covidPlot + geom_point(aes(size=people_vaccinated_est/10^6, color = continent_name, shape = "vacc"), alpha = 1.0)
covidPlot = covidPlot + geom_point(color="black", alpha = 1.0, aes(shape="pop") )
covidPlot = covidPlot + geom_text_repel(hjust=-.2, vjust=.5, size=2.7)
covidPlot = covidPlot + theme_jf_2()
covidPlot = covidPlot + scale_size_continuous(name = translation[["people_1M"]][[lang]],
range =c(1,20),
limits=c(0,max(dfw$population/10^6)) )
covidPlot = covidPlot + scale_color_discrete(name = translation[["continent"]][[lang]])
covidPlot = covidPlot + guides(color = guide_legend(override.aes = list(size = 10)))
covidPlot = covidPlot + scale_shape_manual(name = translation[["pop"]][[lang]],
labels = c(translation[["vacc"]][[lang]], translation[["tot"]][[lang]]),
values = c(16,1),
breaks = c("vacc", "pop"))
covidPlot = covidPlot + guides(shape = guide_legend(override.aes = list(size = 10)))
covidPlot = covidPlot + scale_x_continuous(labels = label_comma())
covidPlot = covidPlot + scale_y_continuous(labels = label_comma())
covidPlot = covidPlot + xlab(translation[["vacc_100"]][[lang]]) + ylab(translation[["vacc_full_100"]][[lang]])
covidPlot = covidPlot + theme(legend.position = "bottom")
return(covidPlot)
}
* Versão inicial. Nesta versão, a população vacinada com ao menos uma dose e a população com vacinação completa é estimada com base nas respectivas medianas da relação entre doses aplicadas e população coberta observada nos demais países.